邵岭教授团队提出息肉分割网络PraNet,计图框架下推理速度大幅提升
计图 (Jittor) 是清华大学自主研发,于2020年3月开源的深度学习框架。Jittor框架因其底层使用了元算子和统一计算图,从而大大提升了深度学习框架的性能和灵活性。最近Jittor医学图像智能分割模型库 JMedSeg的发布,直接为智慧医疗相关领域赋能,促进了研究人员对于算法模型的开发与部署。
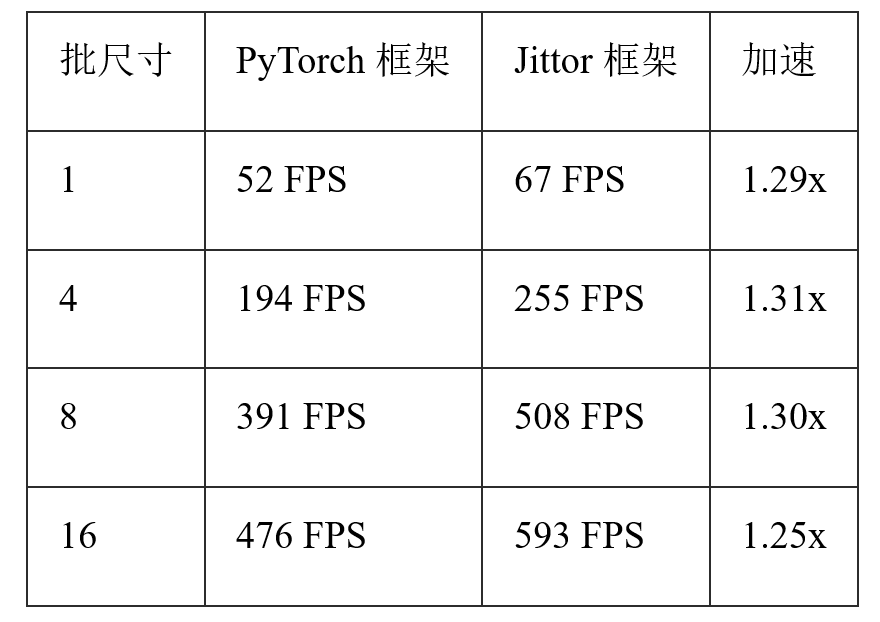
阿联酋起源人工智能研究院(IIAI)邵岭教授团队发表了基于并行反向注意力的息肉分割网络(PraNet),利用并行的部分解码器(PPD)在高级层中聚合特征作为初始引导区域,再使用反向注意模块(RA)挖掘边界线索。实验表明,PraNet优于大多数前沿算法模型并大幅度提高性能,该研究成果发表于MICCAI 2020 [1]。基于Jittor框架下的算法模型相比PyTorch框架,网络推理速度得到大幅提升,当批尺寸为4时,在NVIDIA RTX TITAN显卡上最高达到1.31倍的推理加速。
Part 1
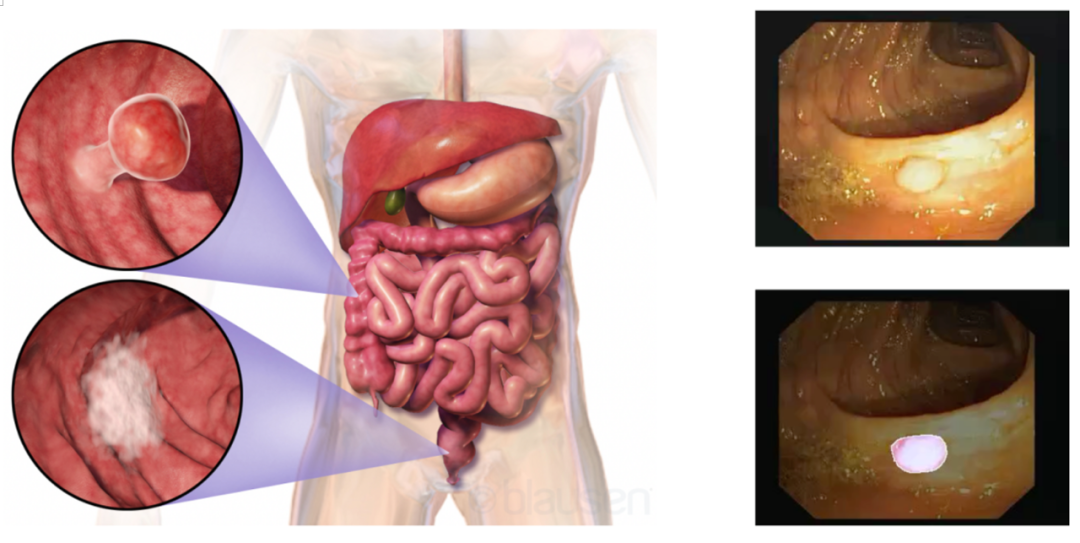
结直肠癌(CRC)是全球第三大最常见的癌症 [2]。因此,通过筛查和切除癌前病变(结直肠腺瘤)来预防结直肠癌至关重要,并已成为世界范围内公共卫生的关注重点。相关研究[3]表明,早期结肠镜检查会使结直肠癌的发病率下降30%左右。因此,在临床实践中对于息肉区域的精确分割是十分重要的。
然而,这是一项具有挑战性的任务,其原因如下:首先,息肉的外观通常不同,例如大小、颜色和质地,即使它们是同一类型的;其次,在结肠镜影像中,息肉与其周围黏膜之间的边界通常是模糊的,往往缺乏通用分割方法中所需的强烈对比度。
Part 2
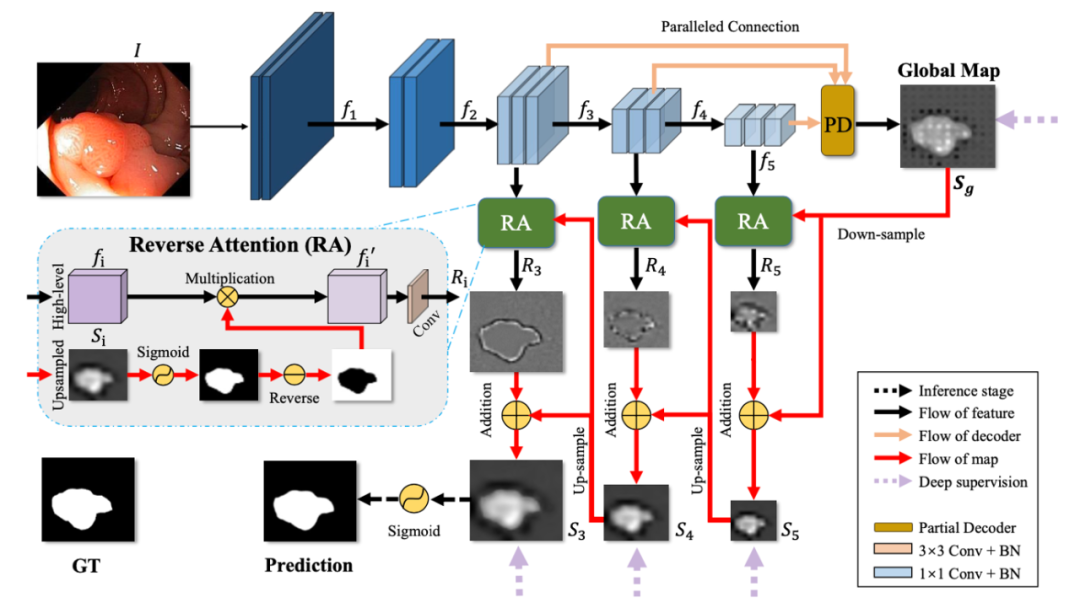
图2 PraNet模型的流程概要
如图2所示,PraNet模型首先使用并行的部分解码器来生成高级语义全局映射图,然后使用一组反向注意力模块来从结肠镜图像中精确分割息肉。每个模块组成部分详细介绍如下:
基于并行的部分解码器的特征聚合:Wu等人在文献[4]中指出,与高层特征相比,低层特征由于其更大的空间分辨率而需要分配更多的计算资源,但对于最终性能的贡献度较小。受此启发,本文使用了并行的部分解码器组件来聚合高级特征,进而得到全局映射图。
反向注意力模块:来自最深层网络所生成的全局映射图只能捕捉息肉组织的相对粗略位置,缺乏结构上的细节。为缓解上述问题,本文提出了一种通过擦除前景对象的方式来逐步挖掘具有鉴别性的息肉区域的策略[5,6]。直观理解,该策略可以通过从高层输出特征中擦除现有的已估计的息肉区域,从而逐步地挖掘互补区域和细节。

Part 3
从表1中可以看到,由于Jittor框架比PyTorch框架更精简且效率更高,在同一张NVIDIA RTX TITAN显卡上,Jittor框架相对于PyTorch框架平均有1.29倍左右的推理加速。特别地,当批尺寸为4时,能达到1.31倍的推理加速,并且计算精度与PyTorch保持一致。
为推动息肉分割这一领域的研究进展,本文所提出模型的Jittor代码、训练/测试数据集和模型评测工具已经全部开源。更多细节请参见如下链接:
https://github.com/DengPingFan/PraNet/tree/master/jittor
参考文献
Deng-Ping Fan, Ge-Peng Ji, Tao Zhou, Geng Chen, Huazhu Fu, Jianbing Shen, Ling Shao, PraNet: Parallel reverse attention network for polyp segmentation, MICCAI.2020, 263-273. Juan Silva, Aymeric Histace, Olivier Romain, Xavier Dray & Bertrand Granado, Toward embedded detection of polyps in wce images for early diagnosis ofcolorectal cancer. IJCARS, 2014, Vol.9, 283-293.
Rawla P, Sunkara T, Barsouk A, Epidemiology of Colorectal cancer epidemiology: incidence, mortality, survival, and riskfactors. Clin Colon Rectal Surg, 2009, 22(4): 191–197.
Zhe Wu, Li Su and Qingming Huang, Cascaded partial decoder for fast and accurate salient object detection. CVPR, 2019, 3907-3916.
Yunchao Wei; Jiashi Feng; Xiaodan Liang; Ming-Ming Cheng; Yao Zhao; Shuicheng Yan, Object region mining with adversarial erasing: A simple classification tosemantic segmentation approach. CVPR, 2017, 6488-6496.
Shuhan Chen, Xiuli Tan, Ben Wang, Xuelong Hu, Reverse attention for salient object detection. ECCV, 2018, 236-252.
您可通过下方二维码,关注清华大学图形学实验室,了解计算机图形学、Jittor深度学习框架、CVMJ期刊和CVM会议的相关资讯。
